In der Exom-Sequenzierung (Whole Exome Sequencing, WES) werden die Protein-kodierenden Bereiche (Exons) aller bekannten Gene, von denen es über 22.000 gibt, analysiert. Geschätzte 85-90 % aller krankheitsverursachenden genetischen Veränderungen sind im Exom zu finden, das etwa zwei Prozent des Genoms ausmacht. Die Exom-Sequenzierung ist damit ein sehr effizientes Verfahren mit hoher Aufklärungsquote. Die Beschränkung der Sequenzierung auf exonische Bereiche birgt aber auch Limitationen. Denn pathogene Varianten machen nicht an Exon-Grenzen halt - unsere Diagnostik aber auch nicht.
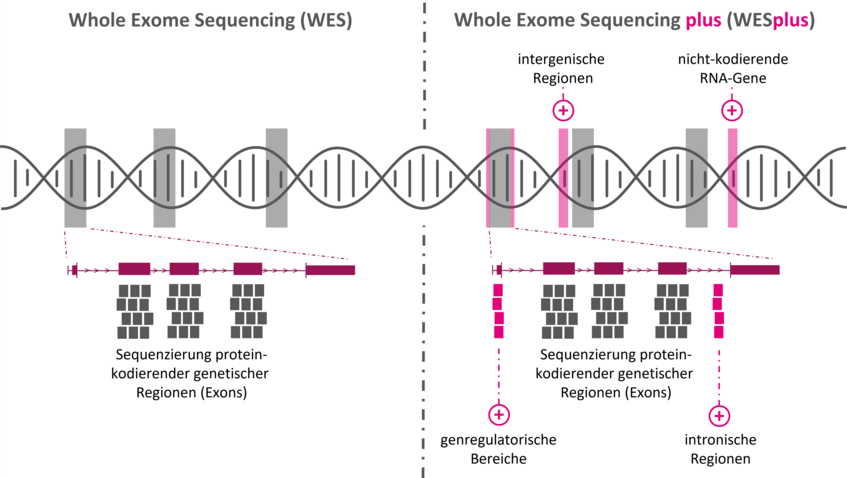
Unsere Stufendiagnostik beginnt mit Whole Exome Sequencing plus (WESplus), einem modifizierten WES-Ansatz, der die Vorteile der Exom-Sequenzierung mit der genomweiten Abdeckung klinisch bedeutsamer Regionen vereint. Für eine sichere Befundung stellt die WESplus Auswertung die klinische Fragestellung in den Fokus und ist maßgeschneidert für Fall und Patient*in. In den seltenen Fällen, in denen die Fragestellung mit WESplus nicht beantwortbar ist, kann als nächster diagnostischer Schritt die Genom-Sequenzierung (Whole Genome Sequencing, WGS) und/oder die Transkriptom-Sequenzierung (RNA-Seq) eingesetzt werden.
Die Basis für WESplus bildet die Exom-Sequenzierung, die gezielt und klinisch-orientiert durch weitere Zielregionen (spike-in Panel) ergänzt wird. Dies ermöglicht eine umfassende Analyse der Gesamtheit aller krankheitsrelevanter genetischen Bereiche. Das spike-in Panel ergänzt das Exom optimal und bildet den aktuellen Wissensstand ab. Dieser ergibt sich aus klinischen Datenbanken (ClinVar, HGMD, PGS Catalogue), den aktuellsten medizinischen und wissenschaftlichen Erkenntnissen sowie unserer in-house Expertise. Bioinformatische Skripte garantieren, dass das Design des spike-in Panels automatisiert aktualisiert wird und so stets up-to-date ist.
WESplus bietet die zielführendste Diagnostik, ist ressourcensparend und kosteneffizient, vor allem aber schafft WESplus einen wichtigen klinischen Mehrwert. Daher gilt: WESplus ist die Basis unserer Diagnostik und nicht ihr Endpunkt.

| |
| |
| |
| |
| |
| |
| |
| |
| |
| |
| |
| |
|
+ umfassende Analyse krankheitsrelevanter Regionen
Zusätzlich zur Analyse protein-kodierender Bereiche (Standard-Exom) sequenzieren wir für unseren WESplus Ansatz genomweit alle bekannten krankheitsassoziierten genetischen Veränderungen. Hierfür greifen wir auf das Wissen klinischer Datenbanken (ClinVar, HGMD, PGS Catalogue), die aktuellsten medizinischen und wissenschaftlichen Erkenntnisse sowie auf unsere langjährige eigene Erfahrung und in-house Ergebnisse zurück.
Klinisch bedeutsame nicht-kodierende, regulatorische und tief-intronische Regionen bilden wir mit hoher Genauigkeit ab. Gene mit besonderer klinischer Relevanz (z.B. PKD1, assoziiert mit der autosomal-dominanten polyzystischen Nierenerkrankung, oder LDLR, assoziiert mit der autosomal-dominanten familiären Hypercholesterinämie) sequenzieren wir in unserem WESplus Ansatz komplett genomisch, d. h. inklusive aller nicht kodierenden, regulatorischen und intronischen Regionen.
Außerdem berücksichtigen wir Einzelnukleotid-Polymorphismen (SNP, single nucleotide polymorphism) mit pharmakogenetischer Relevanz (gemäß CPIC, Clinical Pharmacogenetics Implementation Consortium) sowie über 1.000 SNPs, die die Bestimmung polygener Risikoscores ermöglichen. Hierzu zählen beispielsweise 309 SNPs, die zur Bestimmung des BOADICEA-Riskoscores, mittels dessen das Risiko für eine zukünftige Brustkrebserkrankung eingeschätzt werden kann (https://www.canrisk.org/), bekannt sein müssen. Auch das mitochondriale Genom bilden wir mit WESplus komplett ab.
+ vollständige Abdeckung des mitochondrialen Genoms
Mitochondriale Störungen betreffen viele Erkrankungen. Mutationen im mitochondrialen Genom liegen jedoch häufig nicht in allen Mitochondrien einer Zelle vor. Dieses gemeinsame Vorkommen von Wildtyp- und mutierter mitochondrialer DNA wird als Heteroplasmie bezeichnet. Bei Mitochondriopathien nimmt der Heteroplasmie-Grad (prozentueller Anteil von mutierter zu Wildtyp-DNA) maßgeblich Einfluss auf Schwere und phänotypische Ausprägung der Erkrankung. Bei niedriggrader Heteroplasmie ist es technisch herausfordernd, Varianten sicher zu identifizieren und vom Hintergrundrauschen und technisch bedingten Artefakten abzugrenzen. Wir nutzen einen spezialisierten und optimierten bioinformatischen Workflow, um mitochondriale Varianten sensitiv und präzise zu detektieren. Unser WESplus Ansatz ist für den Nachweis von Veränderungen im mitochondrialen Genom bis zu einem Heteroplasmie-Grad bis zu 2 % validiert.
+ verbesserte und kontrollierte Abdeckung schwieriger Bereiche
Die der Sequenzierung vorgeschaltete Anreicherung der Zielsequenzen haben wir für schwierige Bereiche optimiert. So können wir eine gleichmäßige, (Sequenzier-)lückenlose und hohe Abdeckung für alle Zielregionen gewährleisten und erreichen damit die bestmöglichste diagnostische Sensitivität. Dies erlaubt es uns beispielsweise selbst niedriggradige genetische Mosaike mit einer hohen Wahrscheinlichkeit nachzuweisen.
+ verbesserter Nachweis von Kopienzahlveränderungen
Unser WESplus Ansatz ermöglicht neben der hoch-sensitiven und spezifischen Detektion von Einzelnukleotidveränderungen (single nucleotide variants, SNVs) und von kleineren Insertionen und Deletionen (InDels) ebenfalls den zuverlässigen und hochauflösenden Nachweis von Kopienzahlveränderungen (copy number variants, CNVs). Hierfür kommt eine validierte und hoch-sensitive bioinformatische Berechnung zum Einsatz. So werden Kopienzahlveränderungen nachweisbar, die über die konventionelle Zytogenetik aufgrund der Auflösungsgrenze von zwei bis fünf Millionen Basenpaaren (2-5 Mb) nicht abbildbar sind. Unsere CNV-Analyse wird darüber hinaus durch weitere Sonden ergänzt, um den Nachweis von Mikrodeletions- und Mikroduplikationssyndromen zu ermöglichen. Ohne technischen Mehraufwand kann so in einem einzelnen Ansatz nachgewiesen werden, was bislang nur durch zeit- und ressourcenaufwendige Hybridisierungstechniken wie FISH (fluorescence in situ hybridization) bzw. Array CGH (Array-based Comparative Genomic Hybridization) identifizierbar war. In unserem WESplus Ansatz erreichen wir dabei eine vergleichbare Sensitivität wie das Array CGH-Verfahren.
+ technisch in die Tiefe validiert und hoch-performant
Die Performance und die Sensitivität des WESplus Ansatzes sind gleichwertig mit der eines zielgerichteten Panel-Ansatzes (z.B. eines klinischen Exoms). Dies gilt sowohl für Einzelnukleotidveränderungen und kleine (= unter 10.000 Basenpaare) Insertionen bzw. Deletionen (InDels) als auch für Kopienzahlveränderungen. Durch die gleichmäßige Abdeckung stellt unser WESplus Ansatz zudem eine Alternative zu Array CGH dar.
Durch gezielte Anreicherung und optimierte bioinformatische Tools, die für die Auswertung des WESplus Datensatzes validiert sind, können wir eine hoch-sensitive und hoch-performante Diagnostik selbst für technisch anspruchsvolle und komplexe Regionen gewährleisten.
Vom Design bis zur Datenauswertung erfolgen alle Schritte Leit- und Richtlinien-gerecht und unter Berücksichtigung aller relevanten Datenquellen sowie der aktuellsten medizinischen und wissenschaftlichen Erkenntnisse.
+ Flexibilität in der Auswertung
Die Komplett-Beurteilung des WESplus Datensatzes ist insbesondere bei komplexen, unspezifischen Krankheitsbildern sinnvoll, bei denen eine engere Genauswahl in Form eines Genpanels nicht geeignet erscheint oder die Genpaneldiagnostik bisher nicht zu einer Diagnose geführt hat.
Daneben bietet der WESplus Ansatz auch die Flexibilität, den Datensatz auf das klinische Exom (CES, Clinical Exome Sequencing) bzw. auf Genpanel einzugrenzen. Da dies in silico geschieht, handelt es sich um ‚virtuelle‘ Panels, bzw. ‚virtuelles‘ CES. Für das Panel- bzw. CES-Design greifen wir auf langjährige Erfahrung und Expertise in der indikationsspezifischen Panelsequenzierung zurück. Mittels automatisierter Workflows werden neu beschriebene klinisch relevante Regionen umgehend in das Panel- bzw. CES-Design inkludiert. So kann ganz individuell auf die Fragestellung jeder Patientin und jedes Patienten eingegangen werden – stets unter Berücksichtigung des aktuellen Wissensstandes.
+ zukunftsorientiertes Design
Jedes Jahr werden bis dato unbekannte genomische Regionen mit klinischer Relevanz identifiziert. Unser WESplus Ansatz wird mittels bioinformatischer Skripte automatisiert kontinuierlich an den aktuellsten wissenschaftlichen Kenntnisstand und Updates der klinischen Datenbanken angepasst, um die Erfassung aller relevanten Regionen in einer Analyse zu gewährleisten.
Durch die umfassende Datenerhebung aller exonischen sowie aller bekannten klinisch bedeutsamen Bereiche ist eine Reanalyse der Daten für neu beschriebene Regionen jederzeit und ohne erneute labortechnische Sequenzierung möglich. Eine solche Reanalyse kann z.B. bei Verfügbarkeit neuartiger bioinformatischer Tools und bei neuer Literaturlage sinnvoll sein.
Der WESplus Ansatz enthält nahezu alles, was auf Basis aktueller Literatur und dem gegenwärtigen Wissensstand auswertbar ist. Die Analyse dieses ‚Interpretoms‘ ist sehr anspruchsvoll und setzt eine hohe Expertise des Labors und der wissenschaftlichen Genetiker*innen voraus.
Wir verwenden präzise abgestimmte leitliniengerechte Auswertealgorithmen, die eine hohe Sensitivität im Nachweis und detaillierte Interpretation ursächlicher Varianten garantieren. Eine Klassifikation der Variante erfolgt entsprechend der aktuellsten Leitlinien (ACMG/ACGS/ClinGen). In diesen wichtigen und komplexen Prozess beziehen wir alle relevanten Datenquellen und die aktuellsten medizinischen und wissenschaftlichen Erkenntnisse mit ein.
Eine essenzielle Grundlage für die Dateninterpretation liefern detaillierte klinische Informationen, die entscheidend für eine fokussierte Datenauswertung sind und unbedingt auf dem Anforderungsschein zur molekulargenetischen Analyse vermerkt oder gerne auch in Form von Arztbriefen mitgesendet werden sollten. Je detaillierter die klinische und familiäre Vorgeschichte des Patienten, desto gezielter können relevante Varianten identifiziert werden.
WESplus Analysen können auch vergleichend als Trio-Analysen durchgeführt werden. Hier wird das Erbgut der/des Betroffenen und beider Eltern auf Veränderungen analysiert. Dieser vergleichende Ansatz hat den Vorteil, dass eine direktere Identifizierung neu entstandener (de novo) Varianten oder Aufschluss über den Status der Trägerschaft ermöglicht wird.
Neben der Komplettbeurteilung eines WESplus Datensatzes ist auch eine flexible und völlig freie Zusammenstellung von zu beurteilenden Genen innerhalb des Datensatzes möglich (virtuelles Panel bzw. virtuelles klinisches Exom) (siehe auch „Pluspunkt: Flexibilität in der Auswertung“), um ganz individuell auf die Fragestellung einer/eines jeden einzelnen Patient*in eingehen zu können. Hierfür können Sie gerne jederzeit mit uns Kontakt aufnehmen.